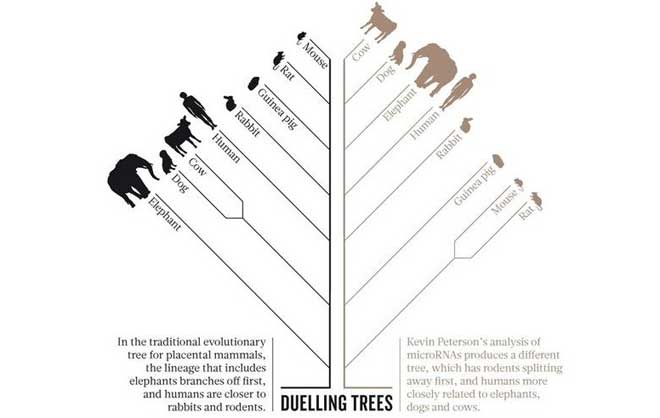
Conforme relatamos aqui e aqui em 2012, o biólogo Kevin Peterson em Dartmouth (na foto à direita) enviou ondas de choque através da comunidade biológia quando estudou os microRNAs e descobriu que eles não rastreavam os relacionamentos evolutivos como esperado. Pensando que os microRNAs deveriam ser janelas claras na evolução, porque eles são geralmente bem conservados, ele continuou “descobrindo problemas desde a base da árvore dos animais até a sua coroa”.
Agora Nature News tem uma atualização sobre a história. A manchete e o subtítulo diziam: “As falhas surgem no método do RNA para construir a árvore da vida: o estudo encontra problemas com a maneira sedutoramente simples de desvendar os relacionamentos evolutivos por meio do microRNA”. Amy Maxmen coloca as más notícias no começo:
Moléculas minúsculas que pareciam fornecer uma maneira poderosa de construir a árvore da vida podem não ter uma capacidade tão forte. Uma equipe de cientistas expôs falhas em um método previamente celebrado que usa moléculas chamadas microRNAs para deduzir relações evolutivas entre animais.
Além de lançar dúvidas sobre alguns resultados específicos publicados nos últimos anos – por exemplo, que as tartarugas estão mais relacionadas com os lagartos do que com pássaros e jacarés – as últimas descobertas derramam água fria no que parecia ser uma abordagem entusiasmante para resolver grandes mistérios na biologia evolutiva . (Enfase adicionada.)
O artigo oferece algumas razões pelas quais houve um grande descompasso. Robert C. Thomson, da Universidade do Havaí em Manoa, por exemplo, questionou porque suas árvores filogenéticas baseadas em sequências genéticas eram tão diferentes das árvores de microRNAs feitas por Peterson. Reconstruindo seus próprios conjuntos de dados, ele descobriu que havia uma forte tendência de os microRNAs abandonarem com o tempo: “este resultado pareceu violar uma das principais justificativas para o uso de microRNAs para construir árvores evolutivas: a ideia de que ele quase sempre é conservado através das gerações”.
Thomson re-analisou conjuntos de dados em microRNAs e encontrou muito mais perdas de microRNA do que o esperado (veja o artigo PNAS). “A forma como os microRNAs estavam sendo analisados provavelmente não refletem a maneira como eles evoluem“, ele acredita agora. Ao ajustar o método, ele obteve melhores resultados:
Thomson e seus co-autores também aplicaram um método alternativo de análise de árvores, que acomodou nuances na evolução de microRNA, em vez de usar um método que pressupõe a conservação de microRNAs através das gerações. Três das cinco árvores resultantes, incluindo a árvore dos répteis, combinaram mais com as produzidas por outros métodos.
Três de cinco é bem próximo da ambiguidade, especialmente usando um método baseado em “nuances na evolução do microRNA”. Por exemplo, Thomson decidiu que a análise bayesiana é mais adequada para avaliar dados de microRNA do que a parcimônia, usada por Peterson. Ele também alegou que houve erro de amostragem na análise de Peterson. Tais questões podem ser questões de opinião; sem a resposta de Peterson, não podemos julgar quais suposições devem prevalecer.
De qualquer forma, se Thomson tivesse resolvido os problemas, seu trabalho de PNAS teria sido uma justificativa para as árvores evolucionárias padrão. Em vez disso, seu objetivo era advertir os outros de usar microRNAs ou qualquer outro método como uma “bala de prata” para resolver relacionamentos.
Thomson oferece outra possível razão para o descompasso: “A discrepância provavelmente resulta do fato de que alguns pedaços de RNA são expressos apenas em momentos específicos da vida de um animal, enquanto os genes no genoma são estáveis”. Essa é uma hipótese testável para explicar as perdas, mas confirmará as árvores filogenéticas? Parece que estudos mais detalhados serão necessários para responder a isso.
A Nature News aponta que Peterson não estava disponível para comentar, mas seu colega Erik Sperling está tendendo a concordar com Thomson, agora acreditando que “MicroRNAs não são a panaceia que talvez esperávamos originalmente”. Peterson e Sperling publicaram um novo artigo usando mais comparações do que apenas microRNAs. Ele colocou tartarugas de volta onde elas pertencem na árvore evolucionária, para o alívio dos darwinistas.
Ainda assim, se isso tivesse minado a bagunça que Peterson criou, a Nature News não teria terminado com uma nota desesperadora:
Para Ken Halanych, um biólogo evolucionário da Universidade de Auburn, no Alabama, o artigo de hoje fornece uma análise crítica de um método que ele duvida há muito tempo. Por que os microRNAs receberam tanta atenção? “Porque estamos esperançosos”, diz ele. Uma ferramenta simples para decodificar como os animais evoluíram ao longo de centenas de milhões de anos seria certamente agradável – mas parece improvável que exista.
Na melhor das hipóteses, isso “explica” a incompatibilidade de microRNA. Mas se uma “ferramenta simples para decodificar como os animais evoluíram” não existe, os evolucionistas ficam com uma coleção de métodos que ou não concordam uns com os outros, ou confiam em fatores “embuste” ad hoc para colocar os dados em acordo com as expectativas. Isso dificilmente é uma boa situação para argumentar a confiabilidade empírica de qualquer teoria científica.
Nosso relato anterior não sustentou, é claro, que os padrões de microRNA suportam o design inteligente, como se cada animal fosse projetado com microRNAs específicos para suas necessidades. Encontrar padrões de microRNA que rastreiam aproximadamente categorias taxonômicas seria esperado, assumindo que cada membro de um táxon tivesse necessidades semelhantes. Estávamos mais interessados em saber como as descobertas de Peterson contradiziam as expectativas darwinianas¹. Nós concluímos anteriormente:
Os defensores do design inteligente não se surpreendem com nada disso, porque seu foco está nas origens da informação funcional, não nas pré-histórias não observáveis, imaginadas assumindo que um ancestral comum universal deu origem a uma árvore genealógica através de mutação aleatória e seleção natural. Os teóricos do design também acreditam em seguir as evidências que levam.
Idem.
Nota do tradutor: o autor anônimo diz que não argumentou a favor do design, mas contra a a origem evolutiva. É certo que caso esses padrões sejam reais, eles podem ser suportados pela hipótese de dependência, mas eu (como programador) ainda creio que as árvores sejam o esquema subjacente de todo sistema informacional, e que neste caso são árvores sobrepostas.
Original: Evolution News. MicroRNAs Still Do Not Support an Evolutionary “Tree of Life”. July 31, 2014. (adaptação)

Faça um comentário